Image: Forma y valor del Genoma
Forma y valor del Genoma
El MNCYT celebra un maratón sobre Biología Molecular
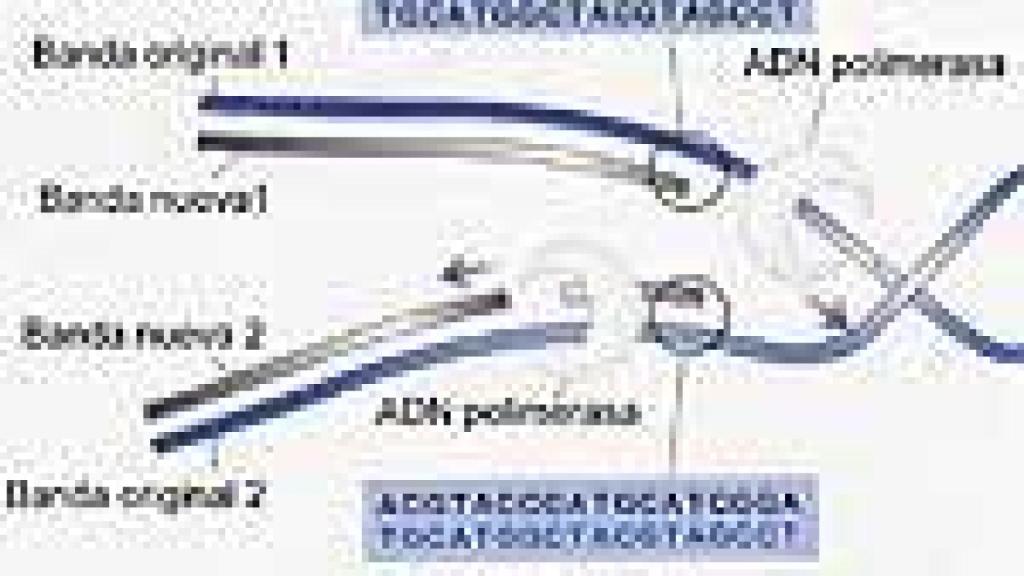
30 enero, 2003 01:00La molécula de DNA replicándose durante el proceso de división celular
Justo cuando se cumplen 50 años del descubrimiento de la estructura del DNA parece necesario reflexionar sobre el vertiginoso desarrollo de la biología molecular. El Museo Nacional de Ciencia y Tecnología de Madrid acoge el 30 de enero el maratón que abordará los desafíos futuros de esta disciplina. Víctor González Huici, del Centro de Biología Molecular, analiza para El Cultural el "nuevo orden" generado con la secuenciación del genoma humano.
El dogma central de la Biología Molecular establece que la información contenida en los genes se transfiere al mRNA (ácido ribonucleico mensajero), y de éste a las proteinas. La expresión de los genes suele estar regulada por proteínas. Así, algunos sólo se expresan durante una etapa concreta de la vida o en determinadas condiciones ambientales y otros lo hacen sólo en ciertos tejidos. Por eso son tan distintas, por ejemplo, una neurona, una célula epitelial o una tumoral. Todas tienen el mismo genoma, pero cada una expresa su propio patrón de proteínas.
El tamaño de los genomas va desde las ~0,6 megabases (Mb) de una bacteria hasta las más de 100.000 de algunos vegetales superiores. El nuestro tiene 3.200 y gracias al Proyecto Genoma Humano se conoce la práctica totalidad de su secuencia.
Homólogos estructurales
Actualmente existen más de 100 genomas totalmente secuenciados, incluyendo los de muchos microorganismos patógenos y los de sistemas modelo como el ratón o la levadura Saccharomyces cerevisiae.
La evolución tiende a conservar las soluciones exitosas incluso en especies muy alejadas, y muchas proteínas humanas tienen homólogos estructurales y funcionales en otros organismos. En general, cuanto más importante es la función de una proteína, mayor es su grado de conservación a lo largo de la escala filogenética. Por todo ello, la secuenciación de un número cada vez mayor de genomas permitirá a la genómica comparativa estudiar sus diferencias y similitudes para descifrar la historia de la evolución a nivel genético, bioquímico y fisiológico. Un primer vistazo a nuestra secuencia genómica revela que tenemos menos genes de los esperados, entre 30.000 y 40.000; esto es sólo unas tres veces más que la mosca de la fruta (Drosophila melanogaster). únicamente un ~2% del total de nuestro DNA codifica proteínas, y más de la mitad está ocupado por secuencias repetitivas no codificantes.
Variabilidad intraespecífica
Por tanto, la clave de nuestra complejidad no reside en nuestro número de genes, sino en su capacidad de dar lugar a muchas proteínas, bien mediante el procesamiento del mRNA (splicing alternativo) o de la proteína (modificaciones post-traduccionales). Otro dato importante es el de la variabilidad intraespecífica: la secuencia se determina a partir de múltiples muestras, procedentes de personas distintas. Más del 99% de la secuencia es idéntica en todos los casos. El resto es lo que diferencia a los individuos.
Se han localizado casi dos millones de sitios en el genoma donde hay variación de una sola base: son los denominados SNPs (single nucleotide polymorphisms o polimorfismos de bases nucleótidos individuales). Pueden darse tanto en regiones codificantes como no codificantes, y si bien muchos de ellos no tendrán trascendencia en la fisiología celular otros pueden provocar desajustes que produzca o predispongan a una enfermedad determinada.
Y es que la secuenciación del genoma humano abre las puertas a la comprensión de la base genética de la enfermedad. La mayoría de ellas tienen, siquiera parcialmente, un origen genético, aunque en el caso de las más comunes como el cáncer, la diabetes, las enfermedades coronarias o las mentales ésta es compleja y no suele ser atribuible a un único gen. No obstante, sólo entre las monogénicas, como la fibrosis quística y el mal de Huntington, ya se han descrito unas 1.500 asociadas a genes de secuencia conocida, habiéndose identificado unos 2.000 genes involucrados en procesos patológicos. La inmensa mayoría de esta información se ha originado en la última década, y continuará aumentando, ya que se desconoce la función de muchos de los genes identificados. Pero al conocer su secuencia y posición, estos genes pueden ser comparados con otros ya caracterizados, tanto en la secuencia humana como en la de organismos modelo, así como analizar directamente su expresión en distintos tejidos y condiciones utilizando técnicas específicas (microarrays), o, si es el caso, diseñar modelos, por ejemplo ratones transgénicos, con el gen homólogo mutado.
En definitiva, no sólo se trata de caracterizar nuevos genes sino de llevar a cabo un estudio sistemático a gran escala para conocer cómo el DNA y las proteínas interactúan entre ellos y con el entorno para crear sistemas complejos, que es el objetivo de la genómica funcional. El estudio de los SNPs permitirá asociar mutaciones concretas de un gen a un determinado riesgo de enfermedad.
Esto se traducirá en la generalización de pruebas diagnósticas basadas en tests genéticos para conocer la propensión de un individuo a enfermedades, como los disponibles actualmente para el Alzheimer o ciertos tumores, y aplicables en medicina forense para identificación de personas o pruebas de paternidad. Desde el punto de vista terapéutico debemos contar con el impulso de la terapia génica, que permitirá sustituir o complementar un gen "defectuoso" con otro sano, y la farmacogenómica.
La capacidad de predecir la respuesta de una célula con genética conocida permitirá adoptar medidas preventivas para evitar la exposición a factores concretos de riesgo así como crear drogas de diseño, individualizadas para cada paciente; así, por ejemplo, a un enfermo de cáncer se le sometería a pruebas para caracterizar los genes defectuosos y emplear una droga específica para su caso. Hay ya algún ejemplo de estas drogas inteligentes, como el caso del STI-571, dirigida contra la proteína aberrante responsable de la leucemia mielogénica crónica. Otra ventaja de los medicamentos del futuro será la reducción de sus efectos secundarios, al poderse conocer la "capacidad genética" de cada paciente para asimilarlos.
Genómica microbiana
Por otro lado, la secuenciación del genoma de animales y plantas de interés comercial permitirá crear especímenes más resistentes y productivos. En cuanto a la genómica microbiana, muchísimo más simple, abre un amplio abanico de aplicaciones sanitarias, industriales y medioambientales: determinación de la base molecular de la patogenicidad y diseño de nuevas drogas y vacunas, tratamiento de residuos, detección de contaminantes, etc…
En definitiva, el desciframiento del genoma humano y de otras especies no sólo supone una gran aportación al conocimiento; el uso inteligente de este enorme banco de datos, procurando los mecanismos adecuados de seguridad y protección de la intimidad, ha de mejorar sustancialmente nuestra calidad de vida en un futuro muy cercano.





