
La investigadora Rebeca García.
Coronavirus: Investigadores de la USC trabajan en el desarrollo de antivirales y vacunas
La investigación del Centro Singular de Investigación en Química Biológica y Materiales Moleculares (CiQUS) ha desarrollado también una aplicación en realidad virtual que permite navegar por las proteínas SARS-CoV-2 en realidad virtual como si fueran una montaña rusa, denominada Corona VRus Coaster
27 julio, 2020 11:11El Centro Singular de Investigación en Química Biológica y Materiales Moleculares (CiQUS) de la Universidade de Santiago (USC) está aplicando la química computacional en la lucha contra el coronavirus, ya que los profesionales de esta institución están trabajando actualmente en el desarrollo de antivirales y vacunas.
La investigadora Rebeca García Fandiño está colaborando, a través de la spin off del CiQUS, MD.USE Innovations, con la empresa americana Ligand Pharmaceuticals Incorporated (NASDAQ: LGND) en la realización de estudios computacionales para ver cómo el Captisol, una ciclodextrina incluida en la formulación del remdesivir que comercializa esta compañía, encapsula el fármaco en el tratamiento del coronavirus.
En este campo de estudio, la investigación del CiQUS liderada por Rebeca García Fandiño, ha desarrollado también una aplicación que permite navegar por las proteínas SARS-CoV-2 en realidad virtual como si fueran una montaña rusa, denominada Corona VRus Coaster. Se trata de líneas de investigación basadas en química computacional que persiguen la transferencia de conocimiento en beneficio de la sociedad.
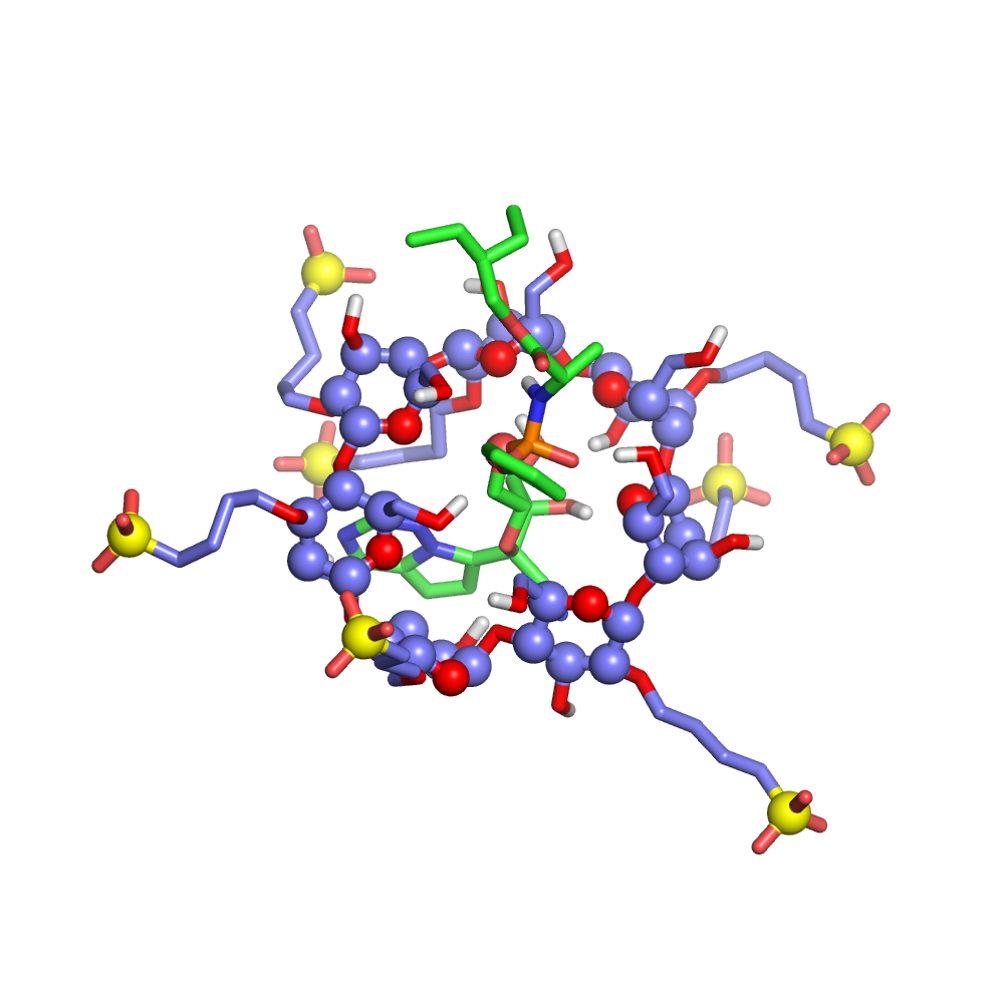
En los estudios computacionales que se están llevando a cabo se utiliza Dinámica Molecular, una técnica que permite simular de manera muy realista cómo se mueve e interacciona el remdesivir con sus excipientes en un ámbito acuoso. Estos “experimentos computacionales” requieren una enorme capacidad de cómputo y se llevan a cabo en centros de cálculo. En este caso, las simulaciones se están realizando en el CESGA (Centro de Supercomputación de Galicia).
Realidad virtual frente al coronavirus
Por su parte, la aplicación Corona VRus Coaster permite navegar a través del esqueleto de 22 estructuras de proteínas involucradas en la infección por el SARS-CoV-2, como si fueran una montaña rusa, proporcionando perspectivas únicas. “La realidad virtual es una herramienta poderosa para profundizar en las estructuras biomoleculares que actualmente se están identificando para la infección por SARS-CoV-2, lo que lleva a avances significativos en la comprensión de la enfermedad y promueve nuevas terapias o tratamientos”, ha explicado la investigadora del CiQUS, Rebeca García Fandiño.
Destaca además que con esta herramienta se busca alimentar una nueva generación de tecnologías inmersivas para la visualización molecular, lo que podría ayudar a comprender y desarrollar un medicamento eficaz contra el SARS-CoV-2, responsable de la pandemia mundial actual.
Software para construir simulaciones de dinámica molecular
Esta línea de investigación de aplicación de la química computacional a la lucha contra el coronavirus se extiende también a otros campos de estudio como la aterosclerosis y otras enfermedades graves del envejecimiento de mano de la farmacéutica Underdog Pharmaceuticals a través de la reciente formalización de un acuerdo de adquisición de la tecnología de la spin-off del CiQUS.
Una colaboración entre ambas entidades que se remonta ya varios años atrás trabajando juntos en la construcción de un nuevo sistema de software, Candymer, que puede construir y parametrizar sofisticadas simulaciones de dinámica molecular de complejos de ciclodextrina-esteroles. Con él, se pueden probar las interacciones de los objetivos farmacológicos y también diseñar moléculas completamente nuevas.





